
2025年3月4日,华南农业大学亚热带农业生物资源保护与利用国家重点实验室、农学院刘耀光/谢先荣团队联合多家单位在Molecular Plant在线发表了题为“SuperDecode: an integrated toolkit for analyzing mutations induced by genome editing”的研究论文。该论文报道了基于Sanger测序、二代测序和三代测序技术对各种基因编辑样品的不同类型突变进行高效检测的超级解码软件工具箱SuperDecode。
研究背景
近年来,基因组编辑技术被广泛应用在基础研究、遗传改良、疾病治疗以及生物技术产业等多个领域,极大地推动了生命科学的发展。然而,随着基因编辑衍生技术和应用场景的不断拓展,所产生的突变类型更加多样化,如何准确、高效地检测基因组编辑结果成为了亟需解决的问题。目前对基因编辑材料的突变类型鉴定,主要是以Sanger测序和二代测序技术为基础进行检测。
刘耀光/谢先荣研究团队前期开发了一套基因编辑突变分析的在线工具DSDecode(Liu et al., 2015; Xie et al., 2017),可以对包含突变靶点的PCR产物测序所获得的峰图文件中直接从重叠的波峰分析出具体的等位突变序列。另外,也有报道基于二代测序混合文库高通量分析基因编辑突变的网络版分析工具如Hi-TOM(Liu et al., 2019)和网络版与本地版CrisprStitch(Han et al., 2024)。然而,在具体的应用过程中,这些工具仍存在一定的局限性。例如,Sanger测序主要适用于二倍体中检测简单的插入/缺失突变,不能检测嵌合突变、低频或复杂类型的突变,且受到通量低的限制,使用Sanger测序对大规模样本检测的成本较高。基于二代测序的突变检测技术提高了样本突变检测的通量,低频率突变和复杂突变的检测能力也高于Sanger测序,但是由于读长短的特点,对基因组大片段的插入、删除以及特定区域内的多靶点编辑引起的结构变异,不能使用二代测序技术进行检测。此外,由于编辑样品的二代测序数据量往往很大,在使用网络版工具通过网络传输大数据时经常遇到不通畅、费时的情况。而使用本地版软件工具可避免大数据传输和数据安全性等问题。
研究内容
为满足科研人员在不同应用场景下的数据分析需求,实现对基因编辑材料的有效和快速鉴定,该团队和合作单位研究人员开发了一款超级解码软件工具箱SuperDecode,包括电脑本地版和在线网络版。SuperDecode可以实现对利用不同测序平台产生的测序数据进行高效分析,并且优化了基于PCR的多样本混合测序文库构建方法,可实现高通量高效数据分析,从而满足科研人员对不同样本通量、不同突变类型鉴定的需求。
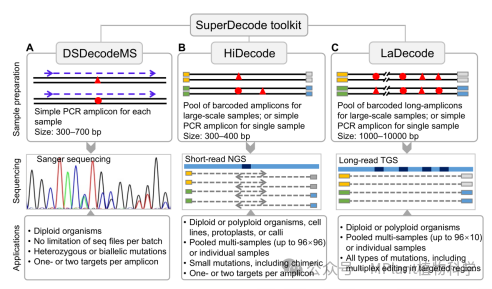
图1 SuperDecode工具箱的分析模块和功能
SuperDecode包含3个子软件模块:DSDecodeMS、HiDecode以及LaDecode,分别对应Sanger测序、二代测序结果以及三代测序数据的解码分析。其中,DSDecodeMS是研究团队此前开发的网页版工具DSDecod/DSDecodeM(Liu et al., 2015; Xie et al., 2017)的升级本地版,添加了去除Sanger测序两端低质量序列的功能,具有更快的分析速度和更为友好的使用界面,可直接读取靶点扩增子的Sanger测序峰图,分析样本的突变类型。HiDecode能对添加特定barcode序列的多样本混合文库的二代测序文件进行自动拆分和突变解码,实现对多种类型样本的高通量分析,包括二倍体、多倍体、细胞系等。HiDecode还提供了96 × n种特异性barcode序列,理论上可一次性检测多达9,216 (96 × 96)个样本的突变类型。LaDecode则是基于第三代单分子测序技术,对包含多个靶点的目的区域进行PCR扩增,并通过添加barcode的方式建立混合文库进行测序和分析。利用三代测序技术读长较长的优势,LaDecode能够识别目的区域中的各种复杂的突变(如靶点之间的片段替换或插入等),并区分检测样本的所有单倍型。因此,该工具特别适用于分析特定区域内进行多靶点编辑(如启动子平铺删除、饱和突变等)引起的复杂变异。该论文提供了利用各模块分析不同基因组编辑样品的示列。
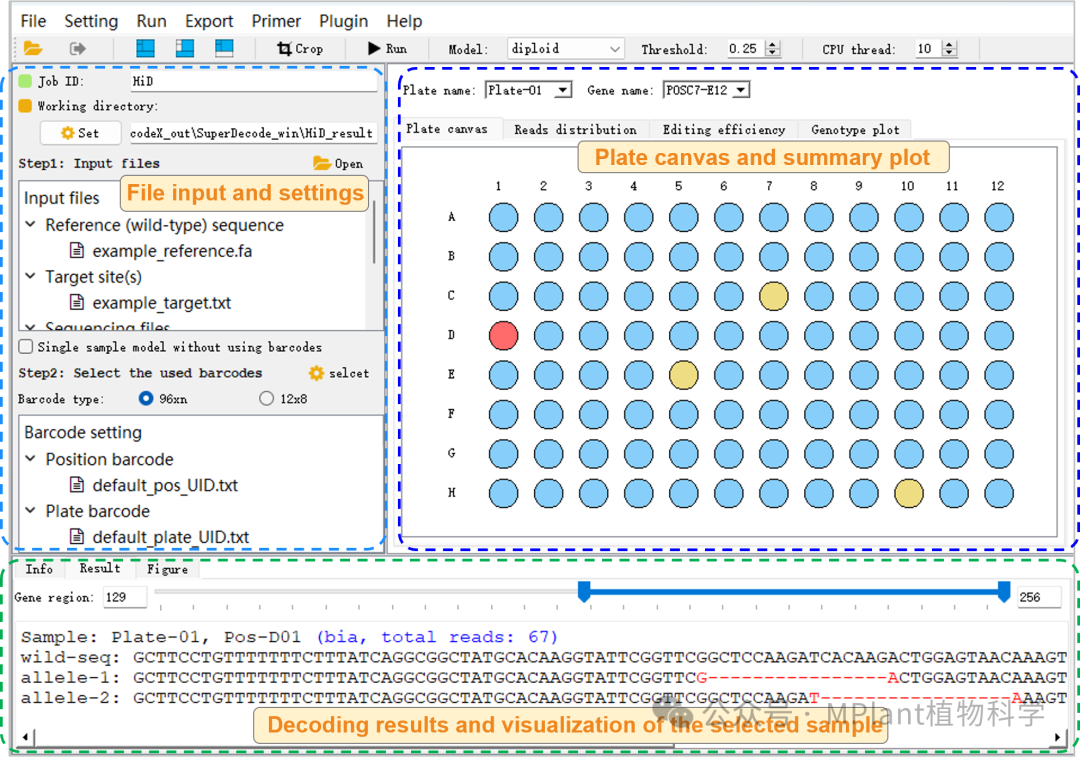
图2 HiDecode模块的可视化操作界面
与已有的相关软件工具相比较,SuperDecode是一个全面、综合的基因编辑突变分析平台,用户可以根据检测样本量、潜在的突变类型特征等选择检测方法和分析模块。SuperDecode具有对用户友好易用的自动化操作界面,使用者不需要具备高专的计算机和生物信息学技能。此外,HiDecode和LaDecode模块支持对用户自定义barcode序列构建的测序文库或者是单样本的测序数据进行分析,具有更加强大的灵活性。除了检测由基因编辑产生的突变,也可以用于检测自然遗传变异,用于基因分型等。
SuperDecode下载
SuperDecode本地版提供了多个版本,包括Windows本地版、MacOS本地版、Linux命令行版。用户可通过代码托管平台GitHub (https://github.com/xiexr/SuperDecode),或奶牛快传(https://tbtools.cowtransfer.com/s/d69cdec128f64e),或者其网络版网页(www.crispr-ge.com/superdecode/) 下载本地版软件包和中文与英文用户使用手册(含详细的建库操作步骤和软件使用方法)。此外,用户也可以通过华南农业大学夏瑞实验室的TBtools-Ⅱ插件商店使用该工具。SuperDecode各模块内置了测试数据包,以帮助用户快速熟悉软件的使用方法。对于测序数据量较小的分析任务,用户可以直接使用SuperDecode网络版(www.crispr-ge.com/superdecode/) 进行在线分析。
作者简介
华南农业大学农学院、广东省高等学校未来作物精准育种基础研究卓越中心、亚热带农业生物资源保护与利用国家重点实验室谢先荣副研究员和刘耀光院士,以及中国热带农业科学院陈程杰副研究员为论文的通讯作者;第一作者为华南农业大学博士生李福权。南京农业大学董小鸥教授,华南农业大学刘向东教授、祝钦泷研究员和吴锦文副研究员,萨斯喀彻温大学马兴亮博士对论文提供了重要的指导和帮助。该项目得到了农业生物育种国家科技重大专项和广东省种业振兴专项的资助。

刘耀光院士团队合影
华南农业大学刘耀光院士团队谢先荣副研究员长期致力于农作物精准育种技术的开发和应用基础研究,从团队课题研究需求出发,开发并分享了一站式植物基因编辑设计和分析平台CRISPR-GE(http://skl.scau.edu.cn/home/)。该平台包含一系列联动工具,如参考基因组/基因序列截取工具seqDownload/geneCat、靶点设计分析工具targetDesign、单碱基编辑靶点设计工具BEtarget、基于微同源介导的片段删除靶点设计MMEJ-KO、基因编辑突变分析工具DSDecodeM等。该平台已为使用基因编辑技术开展基因功能、遗传改良提供了强有力的实用工具与强有力的技术支撑。
参考文献
Liu, W., Xie, X., Ma, X., Li, J., Chen, J., and Liu, Y.-G. (2015). DSDecode: a web-based tool for decoding of sequencing chromatograms for genotyping of targeted mutations. Mol. Plant 8:1431–1433.
Xie, X., Ma, X., Zhu, Q., Zeng, D., Li, G., and Liu, Y.-G. (2017). CRISPR-GE: a convenient software toolkit for CRISPR-based genome editing. Mol. Plant 10:1246–1249.
Liu, Q., Wang, C., Jiao, X., Zhang, H., Song, L., Li, Y., Gao, C., and Wang, K. (2019). Hi-TOM: a platform for high-throughput tracking of mutations induced by CRISPR/Cas systems. Sci. China Life Sci. 62:1–7.
Han, Y., Liu, G., Wu, Y., Bao, Y., Zhang, Y., and Zhang, T. (2024). CrisprStitch: fast evaluation of the efficiency of CRISPR editing systems. Plant Commun. 11:100783.

